
RNA 分離用カラム
- 電気泳動では検出できない不純物の確認が可能
- 分取精製が可能
- 100 ~ 5,000 塩基程度の比較的長い核酸の分離が可能
- 選べる 2 種類の分離モード{逆相・サイズ排除クロマトグラフィー(SEC)}
製品説明
物性
| 充填剤名称 | RNA-RP1 | RNA-SEC-1000 | RNA-SEC-2000 |
|---|---|---|---|
| 分離モード | 逆相クロマトグラフィー | サイズ排除クロマトグラフィー | |
| シリカゲル | 全多孔性球状高純度シリカゲル | ||
| 平均粒子径(µm) | 5 | ||
| 平均細孔径(nm) | - | 100 | 200 |
| 化学結合基 | オクタデシル基 | 親水性基 | |
| 使用可能 pH 範囲 | 2 ~ 7.5 | ||
| 使用圧力(MPa 以下) | 15 | ||
選べる 2 種類の分離モード
コスモシール RNA シリーズには、逆相クロマトグラフィー用カラムである RNA-RP1 と、SEC 用カラムである RNA-SEC-1000 および RNA-SEC-2000 があります。異なる 2 種類の分離モードで、同じ一本鎖 RNA ラダーを分析し、クロマトグラムを比較しました。
逆相クロマトグラフィー用カラム : COSMOSIL RNA-RP1
核酸が長鎖になるにつれて疎水性も増大するため、短鎖と比較して長鎖の核酸のほうが溶出が遅くなります。しかし、配列によって疎水性が異なるため、類似した長さで配列が異なる核酸については、溶出順は鎖長順になるとは限りません。 分析条件を最適化することで、分離を改善することができます。例えば、移動相や温度、グラジエント勾配の設定を変更することができます。※詳細は下記文献をご参照ください。
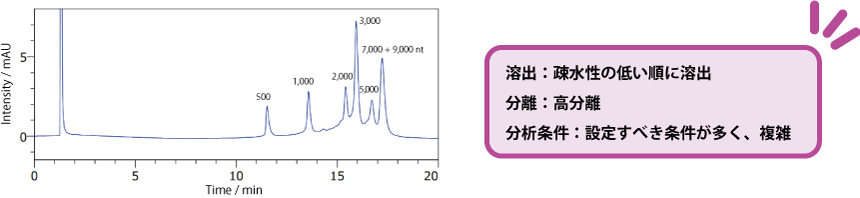
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 4.6 mm I.D. × 100 mm | Temperature | 65℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 8% → 13%(0 → 20 min), 13%(20 → 24 min) |
Detection | UV 260 nm |
| Sample | ssRNA Ladder(New England Biolabs #N0362S) 2.5 times dilution |
||
| Flow rate | 1.0 mL/min | Inj. Vol. | 5 µL |
SEC 用カラム : COSMOSIL RNA-SEC-1000・RNA-SEC-2000
細孔径 100 nm のコスモシール RNA-SEC-1000 と比較して、細孔径 200 nm の RNA-SEC-2000 のほうが、大きな分子の分離に適しています。分離したい分子サイズによって、カラムを使い分けてください。
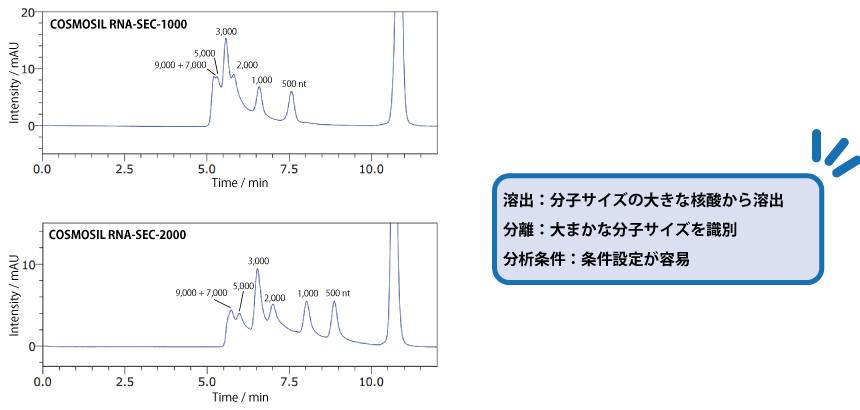
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-**** 4.6 mm I.D. × 250 mm | Detection | UV 260 nm |
| Mobile phase | 20 mmol/L Phosphate Buffer(pH 7.0) | Sample | ssRNA Ladder(New England Biolabs #N0362S) 2.5 times dilution |
| Flow rate | 0.3 mL/min | ||
| Temperature | 65℃ | Inj. Vol. | 2 µL |
充填剤の特長
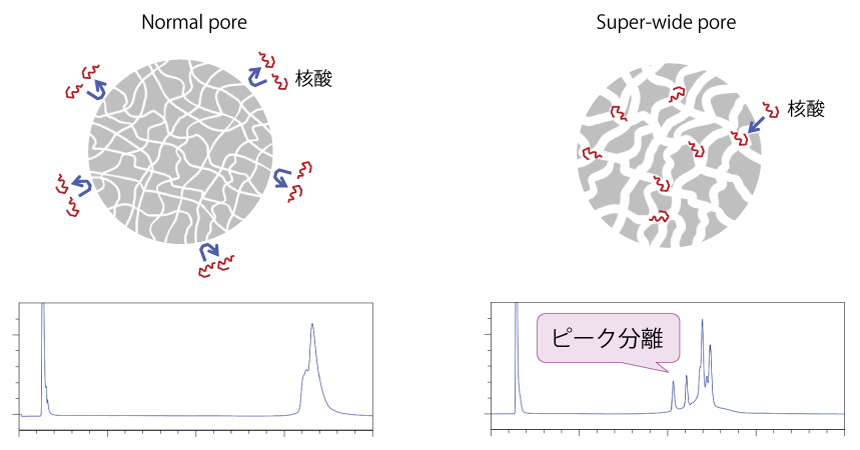
細孔径について
長鎖の核酸分子は分子サイズが非常に大きいため、Normal サイズの細孔には核酸分子が入りづらく、ピークを分離することができません(下図 Normal pore)。Super-wide pore のような大きな細孔であれば、長鎖の核酸分子も内部に入ることができるため、ピークの分離が達成されます(下図 Super-wide pore)。
一般的な HPLC カラムは Normal サイズ(8 ~ 12 nm 程度)の細孔を持つシリカゲルを充填剤として用いていますが、コスモシール RNA-RP1 は Super-wide サイズの細孔を持つシリカゲルを充填剤に用いています。また、RNA-SEC-1000 は 100 nm 、RNA-SEC-2000 は 200 nmの大きな平均細孔径を持つシリカゲルを充填剤に用いていますので、長鎖の核酸分子が細孔に入り、分離することができます。
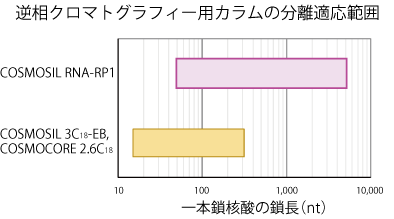
適応分子サイズについて
コスモシール RNA シリーズの逆相クロマトグラフィー用カラム、SEC 用カラムの分離適応範囲を示しました。
※本グラフは概要図であり、必ずしもこの範囲内の核酸分子が分離できるということを示すものではありません。
| 逆相クロマトグラフィー | サイズ排除クロマトグラフィー |
|---|---|
 |
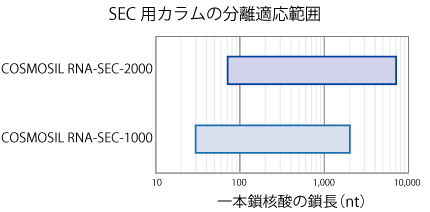 |
| 100 nt を超えるような中長鎖核酸については、細孔径の大きな Super-wide pore シリカゲルを充填剤基剤に用いているコスモシール RNA-RP1 での分析をお薦めします。なお、オリゴ核酸については、充填剤の平均細孔径が小さい 3C18-EB(平均細孔径 12 nm)、コスモコア 2.6C18(平均細孔径 9 nm)での分析をお薦めします。 | コスモシール RNA-SEC-1000 では、100 nt 以下の比較的短鎖の核酸から数千 nt 程度の長鎖核酸まで分離が可能です。 RNA-SEC-2000 では、5,000 nt を超えるような長い核酸の分離も可能です。 |
分析時の注意点
核酸はリン酸基を有するため、ステンレスカラムの内壁などに吸着する可能性があります。弊社では改善策として、リン酸緩衝液入りの移動相{例 : 100 mmol/L Triethylamine Acetate + 20 mmol/L Phosphate Buffer(pH 7.0)}の使用、あるいは、リン酸緩衝液{例 : 20 mmol/L Phosphate Buffer(pH 7.0)}を通液するプレコンディショニングを提案しています。分析時には吸着の有無を確認するため、まず標準サンプルを分析し、分離パターンやピーク形状が安定することを確認してから、実サンプルを分析してください。なお、リン酸緩衝液は不揮発性の塩を含むため、MS 検出器には接続しないでください。
特記事項
このたび、COVID-19 ワクチンに使用されている RNA と同程度の長さの長鎖核酸をコスモシール RNA-RP1 カラムで分析した実験手法の論文が「Analytical Sciences」誌に掲載され、"Hot Article 2023" に選ばれました。
論文名 :
Separation of long-stranded RNAs by RP-HPLC using an octadecyl-based column with super-wide pores
著者名 :
Tomomi Kuwayama, Makoto Ozaki, Motoshi Shimotsuma, Tsunehisa Hirose
誌 名 :
Analytical Sciences DOI : https://doi.org/10.1007/s44211-022-00253-w
移動相や分析温度、グラジエントの条件など、長鎖 RNA の逆相 HPLC 分析を実施するにあたっての情報が掲載されています。ぜひご覧ください。
製品使用例
長鎖 ssRNA の分取精製
逆相クロマトグラフィー用カラム : COSMOSIL RNA-RP1
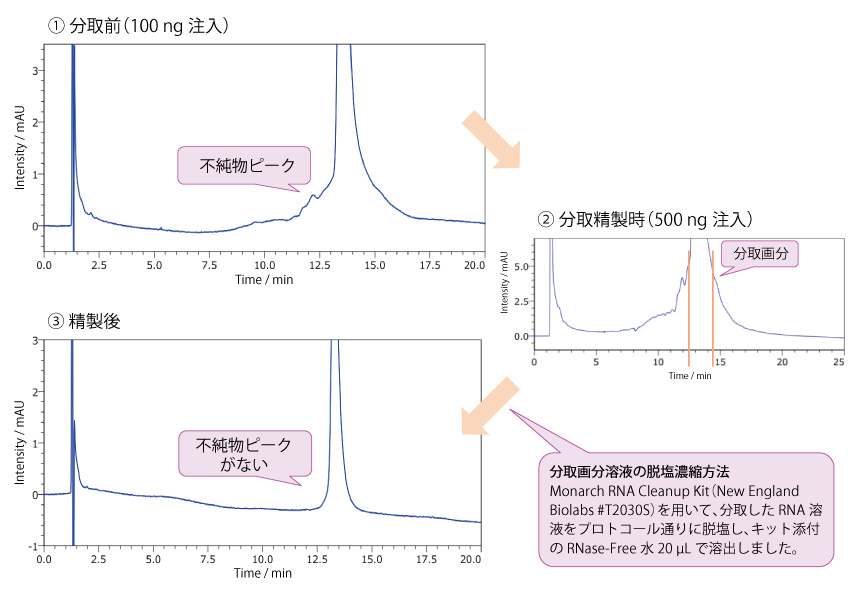
本分析には、in vitro 転写合成した長鎖 ssRNA(4.8 knt)に塩化リチウム沈殿を行ったものをサンプルとして用いました。長鎖 ssRNA をアガロース電気泳動で分析したところ、不純物は確認されませんでしたが{下記分析例(COSMOSIL RNA-RP1)不純物分離参照}、コスモシール RNA-RP1 では、鎖長などが異なると考えられるピークが確認されました(① 分取前のクロマトグラム)。HPLC 精製を行うことで、これらの不純物と考えられるピークを除去することができました(③ 精製後のクロマトグラム)。
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Detection | UV 260 nm |
| Sample | pQBI T7-GFP RNA, digested with HindIII and BamHI(4.8 knt) | ||
| Mobile phase | A : 100 mmol/L Triethylamine Acetate + 20 mmol/L Phosphate Buffer(pH 7.0) B : Methanol / Acetonitrile = 50 / 50 B conc. 11% → 13%(0 → 20 min), 13%(20 → 24 min) |
Inj. Vol. | ① Before preparation(分取前); 100 ng/µL × 1 µL ② Preparation(分取精製時); 100 ng/µL × 5 µL ③ Preparated sample analysis(精製後); desalted solution(undiluted) × 2 µL |
| Flow rate | 0.2 mL/min | ||
| Temperature | 60℃ | ||
各種条件について
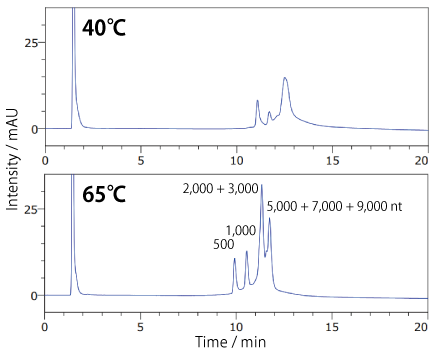
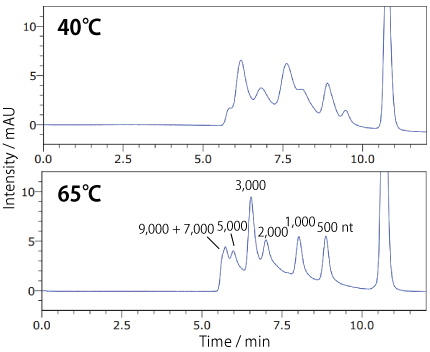
長鎖の一本鎖核酸は低温では高次構造を取りやすいため、ピークがブロードになることがありますが、高温で分析するとピーク形状が改善する場合があります。
分析温度の検討 : 一本鎖核酸逆相クロマトグラフィー用カラム : COSMOSIL RNA-RP1
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Temperature | **℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 7.5% → 17.5%(0 → 20 min), 17.5%(20 → 24 min) |
Detection | UV 260 nm |
| Sample | ssRNA Ladder(New England Biolabs #N0362S) 2.5 times dilution × 2 µL |
||
| Flow rate | 0.2 mL/min | ||
分析温度の検討 : 一本鎖核酸SEC 用カラム : COSMOSIL RNA-SEC-2000
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-2000 4.6 mm I.D. × 250 mm | Detection | UV 260 nm |
| Mobile phase | 20 mmol/L Phosphate Buffer(pH 7.0) | Sample | ssRNA Ladder(New England Biolabs #N0362S) 2.5 times dilution × 2 µL |
| Flow rate | 0.3 mL/min | ||
| Temperature | **℃ | ||
分析温度の検討 : 二本鎖核酸逆相クロマトグラフィー用カラム : COSMOSIL RNA-RP1
非変性状態を維持したまま分析する場合、二本鎖核酸は高温では解離が生じるため、温度条件の最適化が必要です。 二本鎖核酸が一本鎖に解離する付近の温度では、分析温度が 5℃ 異なると、クロマトグラムがダイナミックに変化します。 ※詳細は下記下記文献文献をご参照ください。
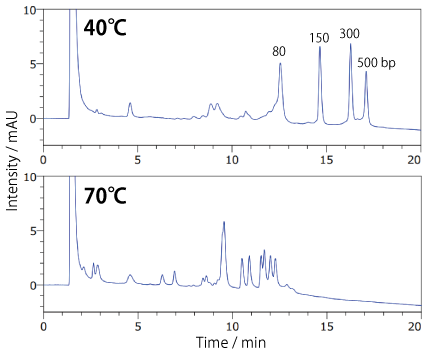
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Temperature | **℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 5% → 20%(0 → 20 min), 20%(20 → 24 min) |
Detection | UV 260 nm |
| Sample | dsRNA Ladder(New England Biolabs #N0363S) 2.5 times dilution × 2 µL |
||
| Flow rate | 0.2 mL/min | ||
分析温度の検討 : 一本鎖核酸 + 二本鎖核酸逆相クロマトグラフィー用カラム : COSMOSIL RNA-RP1
一本鎖核酸が高次構造を取らず、かつ二本鎖核酸が解離しない温度で分析すると、一本鎖核酸と二本鎖核酸を同時に分析することができます。
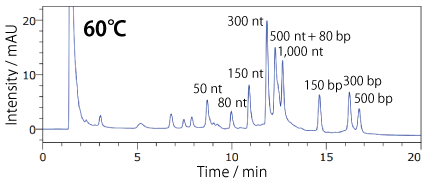
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Detection | UV 260 nm |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 5% → 20%(0 → 20 min), 20%(20 → 24 min) |
Sample | dsRNA Ladder(New England Biolabs #N0363S) 2.5 times dilution × 2 µL + Low Range ssRNA Ladder(New England Biolabs #N0364S) 2.5 times dilution × 2 µL |
| Flow rate | 0.2 mL/min | ||
| Temperature | 60℃ | ||
負荷量の検討 : SEC 用カラム : COSMOSIL RNA-SEC-1000
サンプルを 40 µL 注入しても、2 µL 注入時と同様のクロマトグラムが得られました(縦軸の縮尺は異なります)。
※負荷量はサンプルによって異なります。必ず予備実験を行って注入量を決定してください。
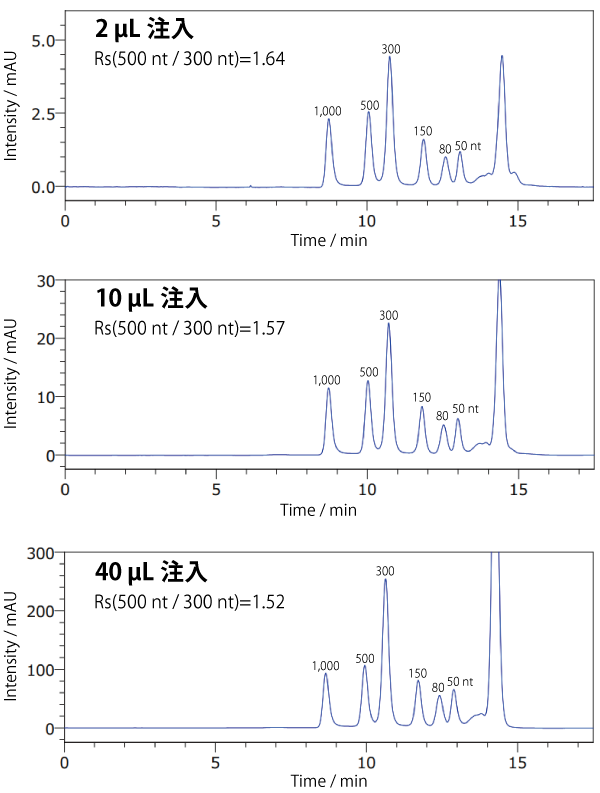
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-1000 7.5 mm I.D. × 300 mm | Temperature | 65℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Flow rate | 1.0 mL/min | Sample | Low Range ssRNA Ladder(New England Biolabs #N0364S) |
分析例 1
COSMOSIL RNA-RP1(RNA 分子量マーカー)Low Range ssRNA Ladder
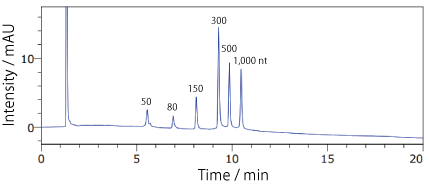
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 4.6 mm I.D. × 100 mm | Temperature | 65℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 5% → 20%(0 → 20 min), 20%(20 → 24 min) |
Detection | UV 260 nm |
| Sample | Low range ssRNA Ladder(New England Biolabs #N0364S) 2.5 times dilution × 5 µL |
||
| Flow rate | 1.0 mL/min | ||
COSMOSIL RNA-RP1(RNA 分子量マーカー)ssRNA Ladder
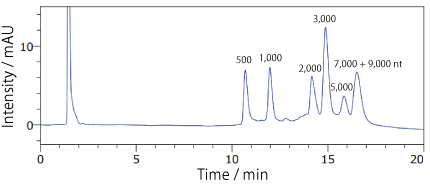
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Temperature | 65℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate +20 mmol/L Phosphate Buffer(pH 7.0) B : Acetonitrile B conc. 7% → 10%(0 → 20 min), 10%(20 → 24 min) |
Detection | UV 260 nm |
| Sample | ssRNA Ladder(New England Biolabs #N0362S) 2.5 times dilution × 2 µL |
||
| Flow rate | 0.2 mL/min | ||
COSMOSIL RNA-RP1(DNA 分子量マーカー)pBR322 / MspI digest
アガロースゲル電気泳動では分離が難しい断片も、コスモシール RNA-RP1 では分離が可能です。
※詳細は下記文献をご参照ください。
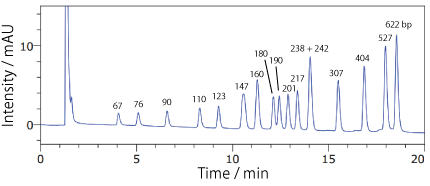
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 4.6 mm I.D. × 100 mm | Temperature | 40℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 10% → 17%(0 → 20 min), 17%(20 → 25 min) |
Detection | UV 260 nm |
| Sample | pBR322 / MspI digest(Nippongene #317-01501) 200 ng/µL × 5 µL |
||
| Flow rate | 1.0 mL/min | ||
COSMOSIL RNA-RP1(DNA 分子量マーカー)100 bp DNA Ladder
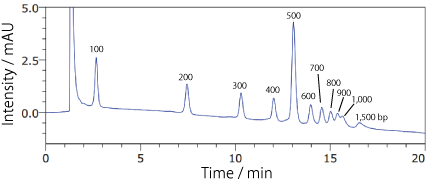
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 4.6 mm I.D. × 100 mm | Temperature | 40℃ |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate(pH 7.0) B : Acetonitrile B conc. 12% → 18%(0 → 20 min), 18%(20 → 25 min) |
Detection | UV 260 nm |
| Sample |
100bp DNA Ladder(Takara Bio #3407A) |
||
| Flow rate | 1.0 mL/min | ||
COSMOSIL RNA-RP1(不純物分離)in vitro 転写で合成した ssRNA
塩化リチウム沈殿により精製した長鎖 ssRNA を分析したところ、アガロースゲル電気泳動では検出されなかった不純物をピークとして確認することができました。in vitro 転写で合成した ssRNA の不純物分離が逆相 HPLC で可能であることが示されています。
※分離したピークの精製については、上記 長鎖 ssRNA の分取精製をご参照ください。
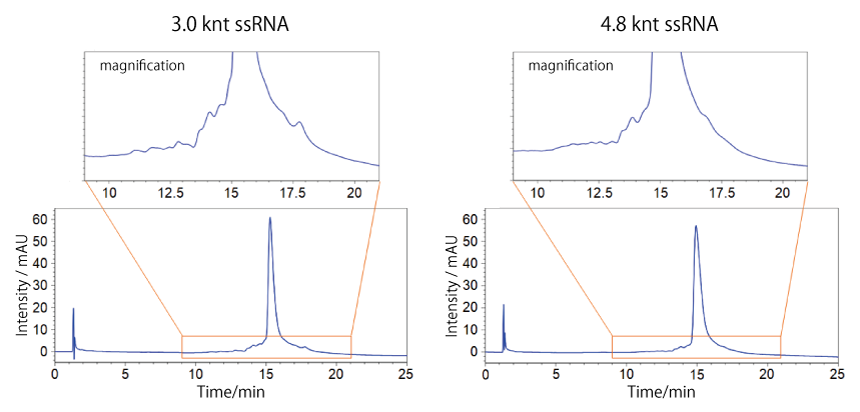
| Conditions | ||
|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm |  |
| Mobile phase | A : 100 mmol/L Triethylamine Acetate + 20 mmol/L Phosphate Buffer(pH 7.0) B : Methanol / Acetonitrile = 50 / 50 B conc. 11% → 13%(0 → 20 min), 13%(20 → 24 min) |
|
| Flow rate | 0.2 mL/min | |
| Temperature | 60℃ | |
| Detection | UV 260 nm | |
| Sample |
pQBI T7-GFP RNA, digested with BglII and EagI(3.0 knt), |
|
分析例 2
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(RNA 分子量マーカー)Low Range ssRNA Ladder(65℃)
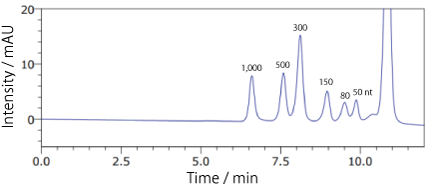
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-1000 4.6 mm I.D. × 250 mm | Temperature | 65℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Sample | Low range ssRNA Ladder(New England Biolabs #N0364S) 2.5 times dilution × 2 µL |
||
| Flow rate | 0.3 mL/min | ||
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(RNA 分子量マーカー)dsRNA Ladder(40℃)
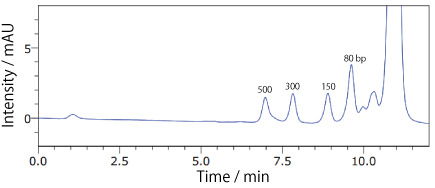
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-1000 4.6 mm I.D. × 250 mm | Temperature | 40℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Sample | sRNA Ladder(New England Biolabs #N0363S) 2.5 times dilution × 2 µL |
||
| Flow rate | 0.3 mL/min | ||
分析例 3
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(DNA 分子量マーカー)ΦX174 / HincII digest
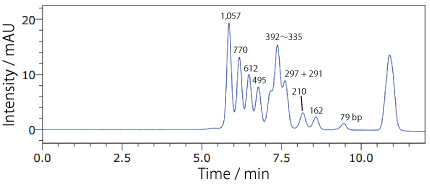
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-1000 4.6 mm I.D. × 250 mm | Temperature | 40℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Sample | X174 / HincII digest(Takara Bio #3406A) 200 ng/µL × 1.25 µL |
||
| Flow rate | 0.3 mL/min | ||
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(DNA 分子量マーカー)20 bp DNA Ladder
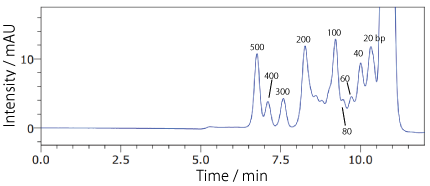
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-1000 4.6 mm I.D. × 250 mm | Temperature | 40℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Sample | 20bp DNA Ladder(Takara Bio #3409A) 200 ng/µL × 2.5 µL |
||
| Flow rate | 0.3 mL/min | ||
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(DNA 分子量マーカー)ΦX174 / HaeIII digest
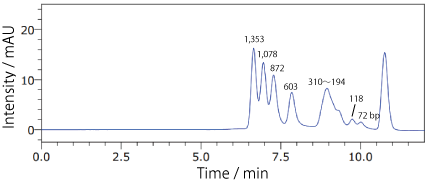
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-2000 4.6 mm I.D. × 250 mm | Temperature | 40℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer (pH 7.0) | Detection | UV 260 nm |
| Sample | ΦX174 / HaeIII digest(Takara Bio #3405A) 200 ng/µL × 1.25 µL |
||
| Flow rate | 0.3 mL/min | ||
COSMOSIL RNA-SEC-1000・RNA-SEC-2000(DNA 分子量マーカー)100 bp DNA Ladder
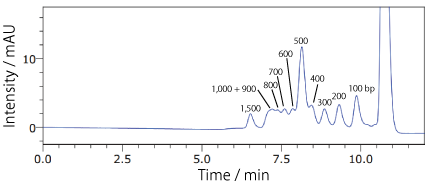
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-SEC-2000 4.6 mm I.D. × 250 mm | Temperature | 40℃ |
| Mobile phase | 20 mmol/L Phosphate Buffer(pH 7.0) | Detection | UV 260 nm |
| Sample | 100bp DNA Ladder(Takara Bio #3407A) 130 ng/µL × 4 µL |
||
| Flow rate | 0.3 mL/min | ||
関連資料
価格表
COSMOSIL RNA-RP1
| 製品名 | サイズ | 製品番号 | オンラインカタログへ |
|---|---|---|---|
| COSMOSIL RNA-RP1 Packed Column | 2.0 mm l.D. × 100 mm | 21078-31 | |
| 4.6 mm l.D. × 100 mm | 21079-21 | ||
| 10 mm l.D. × 100 mm | 21080-81 |
COSMOSIL RNA-SEC-1000
| 製品名 | サイズ | 製品番号 | オンラインカタログへ |
|---|---|---|---|
| COSMOSIL RNA-SEC-1000 Guard Column | 7.5 mm l.D. × 50 mm | 20785-91 | |
| COSMOSIL RNA-SEC-1000 Packed Column | 4.6 mm l.D. × 250 mm | 21088-01 | |
| 7.5 mm l.D. × 300 mm | 19380-21 |
COSMOSIL RNA-SEC-2000
| 製品名 | サイズ | 製品番号 | オンラインカタログへ |
|---|---|---|---|
| COSMOSIL RNA-SEC-2000 Guard Column | 7.5 mm l.D. × 50 mm | 21096-91 | |
| COSMOSIL RNA-SEC-2000 Packed Column | 4.6 mm l.D. × 250 mm | 21095-01 | |
| 7.5 mm l.D. × 300 mm | 19381-11 |
※価格表に記載のないカラムサイズをご要望の際はお問い合わせください。
COSMOSIL / コスモシールはナカライテスク株式会社の登録商標です。
※掲載内容は予告なく変更になる場合があります。


