クロマトグラフィー
合成した長鎖 RNA からの不純物の分離
本ページを PDF でご覧になりたい方はこちら<TN-27>
はじめに
メッセンジャー RNA(mRNA)は、COVID-19 のワクチン開発をはじめ、新しい創薬モダリティとして注目が高まっています。In vitro 転写で合成された mRNA には、製造工程由来の鋳型 DNA や転写酵素のほか、dsRNA といった目的物由来の副産物などのさまざまな不純物が含まれるため、精製が必要になります。従来の精製方法は、除去できる不純物が限定的である、スケールアップが難しい、などいくつかの問題点がありました。それらを解決すべく、長鎖の核酸分離用カラムであるコスモシール RNA-RP1(化学結合基 : オクタデシル基, C18)を用いて、移動相や分析温度などを最適化し、COVID-19 のワクチンと同程度の長さである長鎖核酸中の不純物を分離することに成功しました。本研究成果は、Analytical Sciences. 2023;39(3):417-425. に掲載されています。
長鎖 RNA の分析条件の検討
● 移動相条件の検討
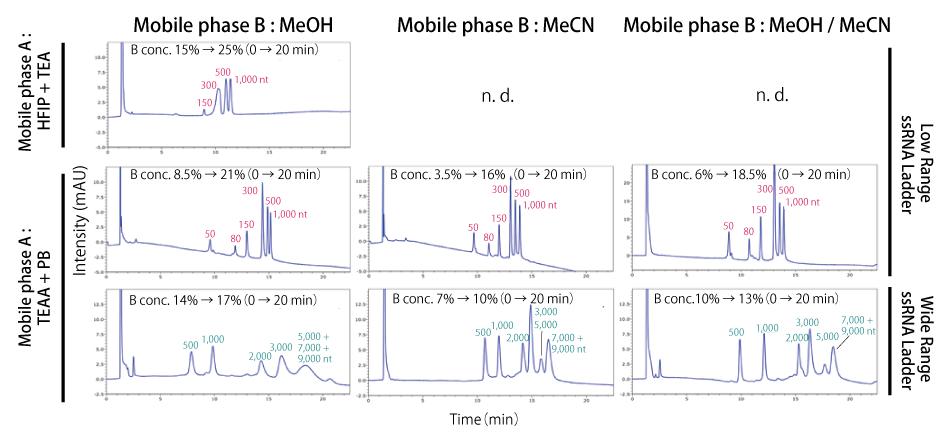
オリゴヌクレオチド(短鎖核酸)の分離に一般的に使用されているイオンペア試薬 HFIP + TEA、TEAA および TEAA に核酸の吸着を抑制するりん酸緩衝液(PB)を添加した 3 種類の移動相を用いて分離条件を検討しました。HFIP を含む場合、アセトニトリルでは溶解できなかったため、メタノールのみで実施しました。
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Flow rate | 0.2 mL/min |
| Mobile phase | A : HFIP + TEA ; 400 mM 1,1,1,3,3,3-Hexafluoro-2-propanol(pH 7.0 with triethylamine) TEAA ; 100 mM Triethylamine Acetate(pH 7.0) TEAA + PB ; 100 mM Triethylamine Acetate + 20 mM Sodium Phosphate Buffer(pH 7.0) B : Methanol or Acetonitrile or Methanol / Acetonitrile(1 / 1, v/v) |
Temperature | 65℃ |
| Detection | UV 260 nm | ||
| Sample | Low range ssRNA ladder(NEB #0364S) Wide range ssRNA ladder(NEB #N0362S) |
||
| Inj. vol. | 2 µL(Sample diluted 2.5 times) | ||
※TEAA の HPLC 結果は TEAA-PB と酷似しているため、論文をご参照ください。
Low range ssRNA において、HFIP + TEA を用いた場合ではピーク形状がブロードになりましたが、TEAA もしくは TEAA + PB を用いた場合ではピーク強度の高い結果が得られました。このことから、長鎖 RNA の分析においては、TEAA もしくは TEAA + PB が適していることが分かりました。続いて、溶媒 B をメタノールからアセトニトリルに変更し、Wide range ssRNA を分析したところ、メタノールを使用すると 50 ~ 300 nt の短鎖 RNA の分離が向上し、アセトニトリルを使用すると 300 ~ 1,000 nt の長鎖 RNA の分離が向上する傾向が見られました。そこで、メタノール / アセトニトリル(1 / 1, v/v)を使用し、分析したところ、短鎖から長鎖まで幅広い鎖長の RNA を分離することができました。このことから、溶媒 B にはメタノール / アセトニトリルが適していることが分かりました。
● 分析温度の検討
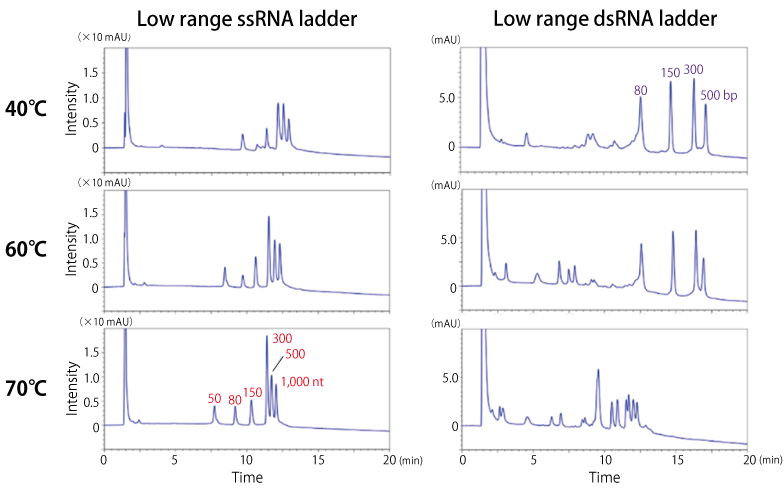
移動相に TEAA およびアセトニトリルを用いた条件で、温度 40℃ ~ 70℃ の範囲で ssRNA および dsRNA を分析しました。
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Detection | UV 260 nm |
| Mobile phase | A : 100 mM TEAA(pH 7.0) B : Acetonitrile B conc. 5% → 20%(0 → 20 min)Linear gradient |
Sample | dsRNA ladder(NEB #0363S) Low range ssRNA ladder(NEB #0364S) |
| Flow rate | 0.2 mL/min | Inj. vol. | 2 µL(Sample diluted 2.5 times) |
Low range ssRNA では、いずれの温度でも分離パターンに差はありませんでしたが、温度を低下させるにつれてピーク強度が減少しました。一方、Low range dsRNA では、40℃ と 60℃ では分離パターンにほとんど差はありませんでしたが、70℃ で分析した際には熱変性により生じた ssRNA と推測されるピークが 9 ~ 12.5 分に確認されました。
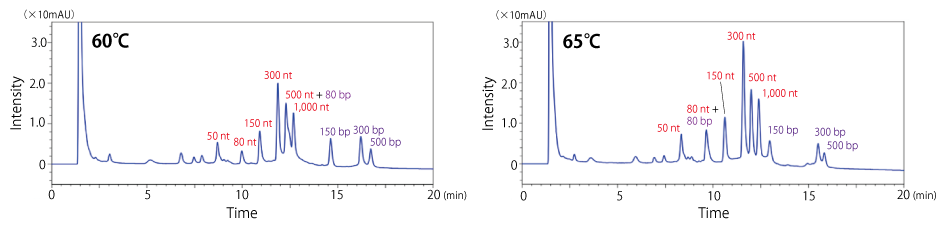
続いて、ssRNA と dsRNA で良好な分離が確認された 60℃ で分析したところ、二本鎖構造を維持しながら ssRNA と dsRNA を分離することができました。一方、65℃ では 80 bp の dsRNA が変性し、80 nt の ssRNA のピークと重なることが確認されました。以上より、ssRNA と dsRNA を非変性状態で分離するには、移動相および温度を精密に制御する必要があることが分かりました。
RNA を取り扱う際の注意点
- RNA は RNase や熱への耐性が低いため、取り扱う際は手袋を着用し、氷上で操作を行ってください。サンプルの希釈や HPLC 装置へのセットの際も同様です。
- RNA はガラスに吸着しやすいため、サンプル溶液は、ガラス製のバイアル瓶ではなくプラスチック製のチューブなどに保存することを推奨します。
- RNA は機械的強度が低いため、過度なピペッティングは避けてください。
分析時の注意点
- 購入した新しいカラムを使用される際は、RNA の吸着を防ぐために、リン酸緩衝液をカラムに十分に通液してください。その後、標準サンプルを分析し、分離パターンやピーク形状が安定していることを確認してから、サンプルを分析してください。
長鎖合成 RNA の不純物分離
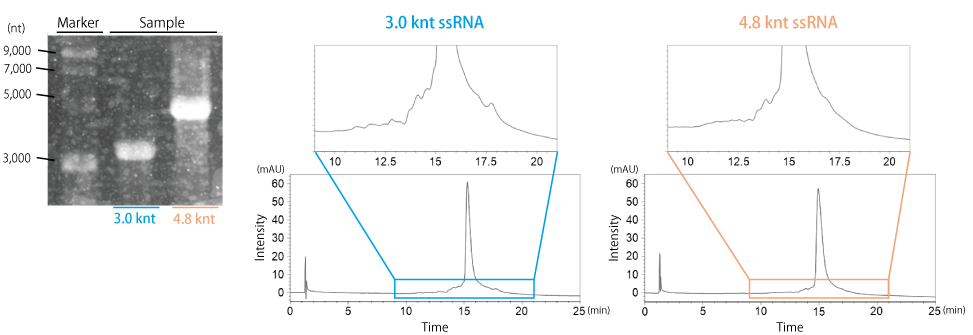
RNA ワクチンで使用されている 3,000 ~ 4,000 nt に近い鎖長の RNA を in vitro 転写で合成し、その不純物分離を試みました。サンプルは、pQBI T7-GFP プラスミドを BglⅡ および EagⅠ で消化した断片および HindⅢ および BamHⅠ で消化した断片より合成した、3.0 knt および 4.8 knt の RNA を用いました。
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 2.0 mm I.D. × 100 mm | Temperature | 60℃ |
| Mobile phase | A : 100 mM TEAA + 20 mM PB(pH 7.0) B : Methanol / Acetonitrile(1 / 1, v/v) B conc. 11% → 13%(0 → 20 min)Linear gradient |
Detection | UV 260 nm |
| Flow rate | 0.2 mL/min | Inj. vol. | 5 µL(100 ng/µL) |
3.0 knt と4.8 knt のどちらの RNA もアガロースゲル電気泳動では不純物を確認できず、高純度に合成されていると考えられましたが、HPLC では主要なピークの他に不純物に由来すると考えられるいくつかのピークが確認できました。これらの結果は、アガロースゲル電気泳動では分離できていない不純物も、HPLC では分離することができたことを示唆しています。
※分取精製の前に、少量のサンプルを用いて、目的物と不純物の溶出位置およびピーク形状が安定することを確認してください。
長鎖合成 RNA の分取精製
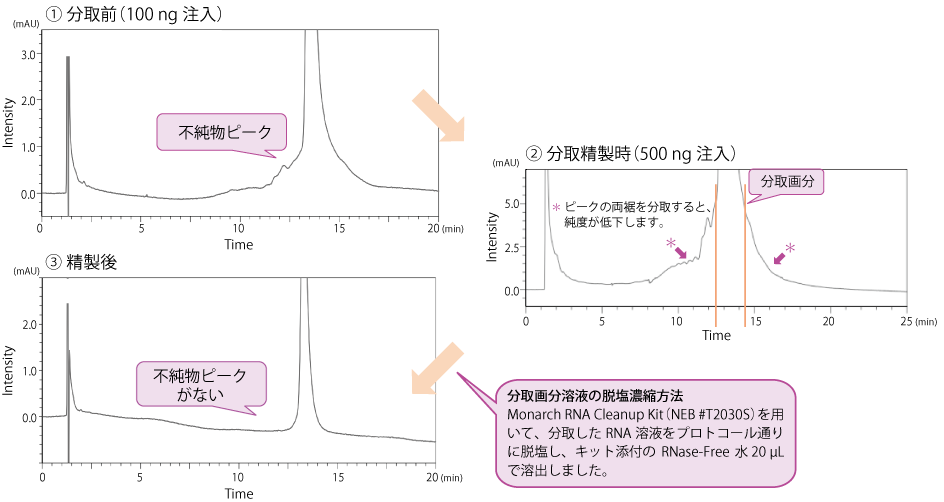
分取精製する際は、サンプルの注入量を徐々に増やしていき(①、②)、目的物と不純物の分離ができ、かつ安定したピーク形状を示す精製条件を確立してください。分取終了後には、不純物が除去できているかの確認を行ってください(③)。
分取精製時の注意点
- 分取精製の際、平衡化時間が短いと分析ごとに溶出時間が変化するため、平衡化時間は 15 分程度とり、時間を短縮しないようにしてください(装置のミキサー容量によって平衡化時間は変化します)。
- 分取する容器はガラス製ではなく、プラスチック製のチューブを使用してください。
- 負荷量はサンプルによって異なりますので、注入量の検討を行ってから精製を実施してください。
その他の分析例
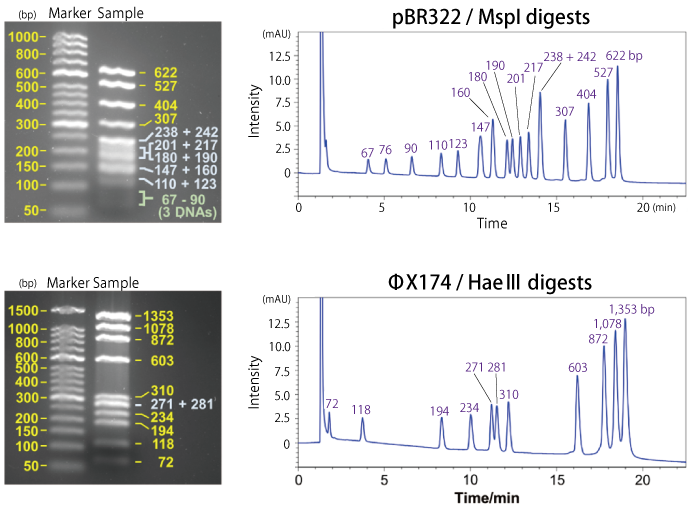
● さまざまな鎖長の dsDNA の分析
コスモシール RNA-RP1 は RNA だけでなく、DNA の分析にも使用することができます。以下に MspI で消化した pBR322 および HaeⅢ で消化した ΦX174 を、それぞれアガロースゲル電気泳動および HPLC で分析しました。
| Conditions | |||
|---|---|---|---|
| Column | COSMOSIL RNA-RP1 4.6 mm I.D. × 100 mm | Flow rate | 1.0 mL/min |
| Mobile phase | A : 100 mM TEAA(pH 7.0) B : Acetonitrile pBR 322 / MspI digests Solvent B conc. 10% → 17%(0 → 20 min) ΦX174 / HaeIII digests Solvent B conc. 12% → 17%(0 → 20 min) |
Temperature | 40℃ |
| Detection | UV 260 nm | ||
HPLC では、pBR322 の MspI 消化産物である 238 bp と 242 bp の断片を除いた全ての鎖長の dsDNA 鎖を分離できました。一方で、アガロースゲル電気泳動では鎖長の近い断片(数 10 bp 以内)はほぼ分離することができませんでした。この結果は、アガロースゲル電気泳動では分離が難しいさまざまな鎖長のサンプルであっても、HPLC であれば分離することができる可能性があることを示しています。また、アガロースゲル電気泳動では短鎖 dsDNA の検出感度が低い傾向にありました。これらのことは合成 RNA の不純物など、アガロースゲル電気泳動で簡易的に検査して確認されない場合でも、HPLC では検出される可能性があることを示しています。
参考文献
- Kuwayama T, Ozaki M, Shimotsuma M, Hirose T. ANAL. SCI. 2023;39:417-425.
DOI : https://doi.org/10.1007/s44211-022-00253-w
※ Selected as HOT Article 2023
関連製品
COSMOSIL / コスモシールはナカライテスク株式会社の登録商標です。
※掲載内容は予告なく変更になる場合があります。
